실험 이론 및 원리
DNA 에서는 exon과 intron이 있으므로 DNA를 template로 하는 경우에는 exon과 intron이 모두 증폭될 수 있다. 하지만 intron은 exon에 비해서 매우 크기 때문에 exon 중에 몇 개를 한꺼번에 증폭하려면 DNA로는 힘들다. 더구나 intron sequence는 일부를 제외하고는 중요성이 떨어진다고 인식되어 밝히지 않은 것이 많아 GenBank에서도 찾을 수 없는 경우가 많다. 이러한 이유 때문에 mRNA를 template로 해서 PCR하는 방법을 RT-PCR(Reverase Transcriptase-Polymerase Chain Reaction)이라고 합니다. 이 경우는 앞에서 설명한 PCR을 시행하기 이전에 RNA로부터 DNA를 합성해 주는 효소(RNA-dependent DNA polymerase)인 reverse transcriptase를 처리한다.
맨 먼저 reverse transcriptase로 RNA를 complementary DNA(cDNA)로 역한다. 이때는 downstream primer만 사용하게 되는데 왜냐하면 RNA는 single strand이기 때문이다. 그 후 생성된 cDNA에서 일반적인 PCR의 과정을 밟아서 유전자를 증폭한다.
downstream primer는 크게 mRNA 뒤에 poly(A) tail이 붙는 것을 응용한 oligo(dT) primer, gene specific primer, 그리고 여기저기 붙는 random primer 이렇게 3가지가 존재한다.
Oligo(dT) primer를 쓰면 분리한 RNA 중 mRNA는 모두 cDNA로 만들어진다. 그리고 Gene specific primer를 사용하면 target gene만 cDNA로 만들어지게 된다. 그리고 random primer를 넣고 반응시키면 길이도 다양하고 모든 RNA(rRNA, tRNA 포함)가 모두 cDNA로 만들어진다. 즉 cDNA pool이 만들어지는 셈이다. Random primer는 잘 안될 것 같지만 뜻밖에도 결과가 좋아서 현재 많이 쓰이는 방법이다. 목적에 따라 다르겠지만, 처음 RT-PCR을 할 때는 우선 random hexamer를 써보고 잘 안되면 다른 것을 시도해 보는게 좋다.
2. Bradford Assay
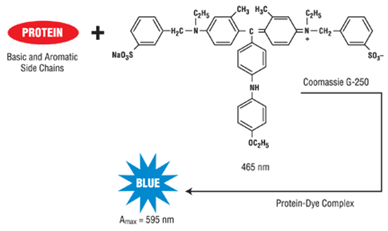
UV-Visible spectrophotometer를 이용, 흡광도를 측정하여 standard물질인 BSA(Bovine Serum Albumin)나 BGG(Bovine Gamma Globulin)를 기준으로 자신의 시료에 단백질이 얼마만큼 들어있는지 측정하는 방법 중 하나이다. 신속하고 간편하게 단백질을 정량할 수 있어 널리 쓰인다. Dye로는 Coomassie Blue G-250를 사용하며, 이 dye가 단백질과 결합해 생기는 파장의 변화를 측정한다.
Acidic enviro㎚ent reagent조건 하에서 단백질은 Coomassie dye에 결합하는데 이 결과로 reddish/brown form(465㎚에서 최대 흡광)이던 dye는 blue form(610㎚에서 최대 흡광)으로 스펙트럼 이동이 일어나게 된다. 두 가지 form의 차이는 595㎚에서 최대가 되는데, 따라서 이 파장이 Coomassie dye-protein complex의 blue form을 측정하는데 최적의 파장이 된다. 575㎚~615㎚사이의 어느 파장에서든 blue form을 측정할 수는 있지만 595㎚에서 측정한 것보다 측정되는 흡광도 값이 10%정도 낮아지게 된다.
Bradford protein assay에서 Coomassie dye ligand는 단백질의 positive charged된 부분에 결합하기 때문에 basic amino acid인 arginine, lysine, histidine잔기가 필요하다. (주로 dye와 작용하는 amino acid는 arginine이다) 또한 Dye-protein complex를 형성하기 위해 van der Waals, hydrophobic interaction도 작용하며, 단백질의 크기 또한 영향을 미친다. 일반적으로 3000Da 미만의 단백질은 Bradford assay로 정량할 수 없다.
Bradford assay는 실온에서 사용가능하며, 어느 특별한 장비를 필요로 하지 않는다. (Spectrophotometer, cuvette, dye정도가 필요하다) 어느 단백질이든 동일한 dye를 더해준 후 약 5분 정도 반응시키고, 595㎚에서 흡광도를 측정하기만 하면 되는 간단한 방법이지만, 대신 Coomassie dye가 염, detergent, 지방 등에 의해 쉬이 영향을 받는다는 단점이 있다.
특히나 sample에 계면 활성제(surfactant)가 조금이라도 존재하는 경우, 정량값이 크게 영향을 받게 되는데, 이는 dye로 사용하는 Coomassie blue가 acidic하기 때문이다. 단백질에 따라 Coomassie dye와 결합하는 정도도 조금씩 다를 수 있으므로 주의가 필요하다. 이런 발색을 이용한 단백질의 정량은 시간이 지날수록 결과값이 달라지는 경우가 생기기 때문에 되도록 빠른 시간내에 시행해야 한다.
단백질의 정량을 위해서는 standard가 필요한데, BSA(Bovine Serum Albumin)나 BGG (Bovine Gamma Globulin)를 이용해 standard curve를 얻을 수 있다. 여러 농도의 BSA or BGG(반드시 0농도를 포함해야 한다)를 만들어 dye를 넣은 후 반응시켜 595㎚에서 흡광도 측정을 하고 그 결과값을 이용해 standard curve를 그린다. 이 때 cureve는 직선형에 가까울수록 신뢰도가 높아진다. 그런 다음 정량하고자 하는 단백질이 든 sample을 dye처리해 흡광도를 측정해 BSA(or BGG)의 값과 비교해보면, BSA는 이미 농도를 알고 있으므로 sample의 흡광도 결과를 이용해 sample의 농도를 계산할 수 있게 된다.
3. SDS page
SDS PAGE(Sodium Dodecyl sulfate-polyacrylamide Gel Electrophoresis)는 1970년에 Laemmili에 의해 개발되었다. 이 방법은 아미노산 측 사슬끼리의 결합(S-S결합 등)을 절단하여 아미노산이 다수 연결된 단일사슬의 polypeptide 상태로 만든다. 이것으로 polypeptide 사슬이 길수록 gel의 망에 걸려 이동도가 작게 되고, 짧은 분자일수록 이동도가 크게 된다.
그러나 단백질은 그것을 구성하는 아미노산 조성의 차이에 따라 각각 특유의 전하를 가지기 때문에, 전압을 걸면 각각의 전하에 따라 이동하게 되고, 단백질의 크기(분자량)의 차이에 의한 분리를 할 수 없다. 거기서 샘플의 단백질의 전하를 똑같이 마이너스로 하기 위해 음이온성 계면활성제인 SDS를 단백질에 결합시켜, 각각의 단백질의 전하를 거의 일정하게 만든 후에 전기영동을 실시한다. 이렇게 함으로써 단백질의 분자량의 차이로 분리하는 것이 가능하게 된다.
즉, SDS-PAGE는 질량의 차이를 이용한 분리방법으로, SDS를 이용하여 모든 단백질이 동일한 (-) 전하를 띄도록 만든 후 질량에 의한 polyacrylamide gel 상에서의 이동속도 차를 이용하여 분리하게 되고, 이 때 sample buffer에 DTT나 mercaptoethanol이 첨가되어 있어 disulfide bond도 끊어지게 되어 단백질은 1차 구조를 형성하여 이동하게 되는 것이다.
4. Genomic DNA
genomic DNA는 긴 이중사슬(107~108)로서 세포의 핵 내에 존재하고 있다. genomic DNA의 추출에 있어서는 어떤 방법으로 파손시키지 않고 추출해서 어떻게 정제도가 높은 것을 얻는지가 중요하며, 이러한 요건을 만족시키는지의 여부에 의해 그 후의 DNA 분석의 성부가 결정된다.
genomic DNA의 추출은 먼저 세포를 용해하고 세포내의 DNA를 가용화한다. 이어서 DNA에 결합하고 있는 히스톤 등의 단백질을 프로테나아제 K라는 단백분해효소를 이용하여 제거한다. 그리고 유기용매인 페놀 클로로포름을 DNA 용액에 첨가하여 DNA 용액 중의 단백질을 변성시켜서 제거한다. 클로로포름은 지질제거에도 효과적이다.
그리고 또 RNA 분해효소인 RNase를 사용하여 DNA 용액 중에 혼재하고 있는 RNA를 분해, 제거한다. 그 후 사기 페놀, 클로로포름 처리한 후 DNA 용액 중에 초산나트륨과 에탄올을 가하여 냉각시키면 DNA는 침전되고(에탄올 침전법) 원심분리하면 pellet으로 회수할 수 있다. 이렇게 DNA를 추출의 조작을 제 단백이라는 것이 된다. 더욱이 DNA 추출을 통해서 DNA에 물리적인 절단이 되지 않도록 하는 것이 중요하다.
실험 기구 및 시약
1) upstream primer, downstream primer, dNTP, 10x PCR buffer,
2) Taq polymerase, distilled water, cDNA
2. Bradford assay
1) BSA(bovin serum albumin), 5X Bio rad protein assay(Bradford 용액)
2) 96well plate, 알고자 하는 protein, microplate reader
3. SDS-page
1) distilled water, 30% acrylamide mix, 1.5M Tris, 10% SDS,
2) 10% ammonium persulfate, TEMED, 1.0M Tris, n-butanol
4. Genomic DNA prep
1) 대장균 암세포, Ethanol, Loading dye, 0.7% Agarose gel, DNase,
2) EN buffer(50mM Tris-HCl, 10mM EDTA, 100mM NaCl, SDS 1%).
3) Sodium acetate, 1cc 주사기




!&emoji=☕&slug=xFu35q9&button_colour=5F7FFF&font_colour=ffffff&font_family=Poppins&outline_colour=000000&coffee_colour=FFDD00)



![[일반생물학실험]온도와 pH가 효소작용에 미치는 영향 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEjdvNA5S925m8lScjFhJj-uy5SdCcaNFG1AuR_7BbQbDEoHBDEEkqfM64sipvro3RydmSO4_MS2794o2bJ0FCljQ31pcD5W39ZGOHROD7qKXLO1rlm2ZuBtbxJ9CZHSnsk2v1KSEsL4EGA/w680/%25EC%2598%25A8%25EB%258F%2584%25EC%2599%2580+pH%25EA%25B0%2580+%25ED%259A%25A8%25EC%2586%258C%25EC%259E%2591%25EC%259A%25A9%25EC%2597%2590+%25EB%25AF%25B8%25EC%25B9%2598%25EB%258A%2594+%25EC%2598%2581%25ED%2596%25A5.PNG)
![[일반생물학실험]여러 조건에 따른 효소 반응](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEj4kaETe9N8j9q9-ClYy9V1kM2L9s5l19K6YoBOE0SB70BmssQXzVADvQijp1ieiQ9mIxcmCwz9e-YCQFVnXYR7m1vmfjTSs0ZNxCmBLG7wgzRBqiY1Kizx8YuhSdw-RDbs1PohokUd69c/w680/%25EC%2597%25AC%25EB%259F%25AC+%25EC%25A1%25B0%25EA%25B1%25B4%25EC%2597%2590+%25EB%2594%25B0%25EB%25A5%25B8+%25ED%259A%25A8%25EC%2586%258C+%25EB%25B0%2598%25EC%259D%2591.PNG)
![[일반생물학실험]항생제 감수성 검사 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEhwCJEjB_r2Ojsrn244q-fQmpHsDyUUfgWMUnsfhPKQeN2e7_u1pN_ZuwIdxPLVWS6LFywJUrDTYWJe2V6ZVVb1PtfyvX62Y15OxPfXoiaJkqVoSir3ZoFAmDpe5GcoMBmxEsJUKsSvWYc/w680/%25ED%2595%25AD%25EC%2583%259D%25EC%25A0%259C+%25EA%25B0%2590%25EC%2588%2598%25EC%2584%25B1+%25EA%25B2%2580%25EC%2582%25AC.PNG)
![[고분자공학실험]PMMA 중합 반응 - MMA, AIBN 정제와 PMMA Bulk 중합 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEj5hkRZhYWcXolP679_yO0kyP8_R-G9dlTqk5AGEdfsIq9M5K3DJc_tvpYck4x1kciBm2IWwFygrzfWH-HPzUjXiCHAFrjv9zeKYDxAl2tGNqgryyc3t9y4B9lQveS_coAqnmZw4pX4Hyo/w680/PMMA+%25EC%25A4%2591%25ED%2595%25A9+%25EB%25B0%2598%25EC%259D%2591.PNG)
![[일반화학실험]산화-환원 적정 : 비타민 C의 정량 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEintFUW1pYHbKK8MPzki4vdqVMjTUEFFxPdZB1sTJQw13XSFZWqcN5w0x1AlyzfErlYYSgoNqBni4YGlpFC4ZeULK9VHbhfIXLiuZ3UWTl2oBRua7FE8UUQqtYEx3498FENAnlHlh0f8RFkT7Na8jveOtBtQ5UbY8jE80ZCwFoU_jqw_0Z6BofRYZF7/w680/%EC%82%B0%ED%99%94-%ED%99%98%EC%9B%90%20%EC%A0%81%EC%A0%95%20-%20%EB%B9%84%ED%83%80%EB%AF%BC%20C%EC%9D%98%20%EC%A0%95%EB%9F%89.PNG)
0 댓글