실험 방법
1) cutting, uncutting DNA 1λ에 10X buffer 5λ, dNTP 2λ, primer(up, down) 1λ씩, Taq polymerase 1λ, d.w 39λ와 DNA를 빼고 d,w만 40λ넣은 negative control을 각각의 PCR용 tube에 넣었다.
2) 기계에 넣고 PCR 시켰다.
3) 일주일 후 1% agarose gel을 만들고 전기영동 시킨 후 자외선 사진 촬영을 했다.
2. Bradford assay
1) 얼려둔 protein을 아이스 박스에서 천천히 녹였다.
2) 5x bio rad protein assay(bradford 용액)을 1/5로 희석했다.
3) 96well plate를 이용하여 BSA 1㎎/㎖을 10배 희석한 0.1㎎/㎖을 0, 2, 4, 6, 8, 10, 20, 50㎕씩 넣고 bradford 용액을 각각의 부피합이 200㎕이 되도록 넣어줬다.
4) protein 원액, protein 1/2 희석액, protein 1/5 희석액, protein 1/10 희석액을 각각 1㎕씩 3번취해서 plate에 넣어주고 마찬가지로 bradford 용액을 각각의 부피합이 200㎕이 되도록 넣어줬다.
5) microplate reader로 630㎚에서 흡광도를 측정하였다.
6) 도출된 흡광도 값으로 단백질의 양을 측정하였다.
3. SDS-page
1) running gel을 만들어서 조립된 유리판 사이에 넣고 n-butanol을 넣었다.
2) running gel이 굳었을 때 n-butanol을 버린 후 stacking gel을 만들어서 running gel 위에 붓고 comb를 꽂았다.
3) stacking gel이 굳는 동안 단백질 sample과 5x loading dye를 넣고 5분간 끓였다.
4) stacking gel이 굳은 후 comb를 뽑은 다음 3번 sample를 loading 시켰다.
5) 60V에서 30분간 전기영동 하였다.
4. Genomic DNA prep
1) plate에 있는 상등액을 버리고 TEN buffer 400㎕을 넣고 섞었다.
2) plate에 있는 액체가 끈적끈적 해졌을 때 그것들을 새로운 1.5㎖ tube에 옮긴 후 에탄올 800㎕와 sodium acetate 40㎕을 넣고 섞었다.
3) pellet을 다른 새로운 1.5㎖ tube에 옮기고 10분간 air dry 시켰다.
4) distilled water 100㎕을 넣고 녹였다.
5) 4번의 DNA에서 4개의 tube를 준비해서 1개에는 DNA 9㎕ + 10X Loading dye 1㎕을 넣고 0.7% agarose gel에 전기영동 시켰다.
6) 또 다른 tube에 25㎕ DNA + DNase I 2㎕+10x Loading dye 3㎕을 넣고 10분간 37℃에 놓고 전기영동 시켰다.
7) 또 다른 tube에 25㎕ DNA + DNase I 10㎕+10x Loading dye 4㎕을 넣고 10분간 37℃에 놓고 전기영동 시켰다.
8) 또 다른 tube에 25㎕ DNA + distilled water 300㎕을 넣고 1cc 주사기로 약 30회 섞고 에탄올 600㎕과 3M Sodium acetate 30㎕을 넣고 centrifuge 한 다음 9㎕를 취해서 10x Loading dye 1㎕을 넣고 spin down 시킨다음 전기영동 시켰다.
9) 자외선 촬영으로 결과를 관찰하였다.
실험 결과
맨 왼쪽 size marker 500~600bp 사이 위치에 밴드들이 보였다. 우리가 원하는 product size(542bp)하고 일치했다.
2. Bradford assay
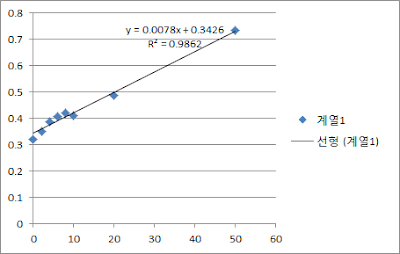 |
| BSA curve |
BSA(0.1㎎/㎖) | absorbance | 시료의 농도 | 측정1 | 측정2 | 측정3 | y | x | x값 × 희석배수 | 단백질20㎍ 취할 때 필요한 시료부피(㎕) |
0 | 0.322 | 원액 | 0.713 | 0.755 | 0.663 | 0.710333 | 47.15 | 4.7145299 | 4.242204496 |
2 | 0.35 | 1/2 희석 | 0.437 | 0.48 | 0.456 | 0.457667 | 14.75 | 2.9504274 | 6.778679027 |
4 | 0.387 | 1/5 희석 | 0.482 | 0.371 | 0.352 | 0.401667 | 7.57 | 3.7863248 | 5.282167043 |
6 | 0.409 | 1/10 희석 | 0.42 | 0.369 | 0.29 | 0.359667 | 2.19 | 2.1880342 | 9.140625 |
8 | 0.422 |
|
|
|
|
|
|
|
|
10 | 0.41 |
|
|
|
|
|
|
|
|
20 | 0.487 |
|
|
|
|
|
|
|
|
50 | 0.733 |
|
|
|
|
|
|
|
|
BSA curve에서 98.6%의 직선적인 관계가 인정되었지만 같은 시료를 썼음에도 불구하고 단백질을 취할 때 필요한 시료의 부피가 균일하게 나오지 않았다.
3. SDS-page
transfer단계까지 안하고 running단계까지만 실험을 하였다. running을 걸 때 기포들이 위로 부글부글 올라가는 걸 관찰하였고 맨 오른쪽에 보이는 것이 size marker이고 왼쪽의 4개 sample에서 단백질 밴드들이 관찰되었다. marker의 정확한 밴드당 dalton을 알 수 없어서 분자량은 측정하지 못하였다.
4. Genomic DNA prep
맨 오른쪽부터 gDNA, gDNA+DNaseI 2㎕. gDNA+DNaseI 10㎕. gDNA+물 300㎕+sonication+Ethanol 600㎕+Sodium acetate 30㎕ 순서이다. 전체적으로 DNA가 밑으로 쭉 끌리는 모양을 얻을수 있었고 sonication을 한 것에서는 위 아래에 진한 밴드가 나뉘어서 관찰되었다. 또한 DNase 2㎕를 처리 했을 때 아무것도 처리 안한것에 비해서 밑으로 좀 더 끌린 걸 볼 수 있었고 10㎕를 처리했을때는 DNA가 거의 분해되서 희미한 밴드 몇 개만 관찰할 수 있었다.
토의 사항
RT-PCR에서는 조원 모두의 sample에서 원하는 product를 얻을 수 있었다. 2,3번째 sample이 희미하게 나온 것은 PCR때 증폭이 덜 된 것으로 RNA에서 cDNA를 만드는 과정에서 RNA가 오염이 되어서 cDNA가 복제가 별로 안되서 그랬을 것이다.
그리고 bradford assay에서 선형성이 약간 떨어지는 것으로 나왔는데 이러한 선형성을 올리기 위해서는 reagent를 넣고 샘플마다 incubation time을 일정하게 유지시켜 주거나 standard의 최대 흡광도보다 sample의 흡광도가 큰지 확인하고 만약 큰 값이 있다면 제외시켜야 한다. 이 외에도 기본적인 pipetting, BSA자체의 문제점이 있을 수도 있다. 이번 실험에서 R^2값이 나온 것은 pipetting 문제 같다. 선형성 뿐만 아니라 같은 시료를 썼음에도 불구하고 원액에서의 x값과 희석액 에서의 x값에 희석 배수를 곱해준 것을 비교 해보면 너무 다르기 때문이다.
그리고 SDS-page에서 단백질을 왜 95℃에서 변성시키는지 추가적으로 한번 조사해봤다. SDS-PAGE 의 과정을 살펴보면 우선 단백질 샘플을 loading buffer와 섞어준 다음 gel에 주입해야 하는데, loading buffer에는 SDS가 포함되어 있다. 이 SDS가 단백질을 둘러싸 micelle을 형성하면 둘러싸인 단백질은 단백질의 사이즈에 비례하는 전하를 가지게 된다.
이 때 사이즈와 둘러싸이는 전하의 양, 즉 SDS의 양을 일치시키려면 꼬여있는 단백질을 일자로 풀어주어야 오차가 적어지게 되는데 단백질이 아주 많이 꼬여있으면 실제 분자량보다 크기가 작아져 SDS가 많이 둘러싸지 못해 원래보다 작은 분자량에서 나타나게 된다. 이러한 이유 때문에 가열을 통해 단백질을 풀어줌으로써 SDS가 단백질의 크기에 비례하여 둘러쌓을 수 있게 해주는 것이다.
Genomic DNA 분리에서는 gel을 옮기다가 떨어뜨려서 gel이 망가지는 바람에 샘플을 다양하게 관찰할 수 없었다. 전체적으로 쭉 늘어진 모양을 관찰할 수 있었는데 이것은 각기 다른 size의 DNA들이 heterogeneous하게 섞여 있고, prep과정에서 약간의 DNA가 깨졌기 때문이다. 게다가 실험을 할 때 Genomic DNA가 너무 많이 뽑혀버렸기 때문에 아무것도 처리 안한 sample에서 band가 굉장히 두껍게 관찰되었다.
그리고 DNase를 2㎕처리한 샘플에서는 DNA가 아무 처리를 안한 것 보다 훨씬 더 쭉 늘어진 상태를 볼 수 있었는데 이것은 여러종류의 DNA가 DNase에 의해 불규칙적으로 잘리면서 다양한 길이의 DNA 조각들이 생겼기 때문이다. DNase를 10㎕처리한 sample에서는 너무 많은 DNase를 넣어서 DNA가 완전히 분해 되버려서 희미한 밴드 하나만 관찰이 되었다. 그리고 sonication을 한 sample에서는 물리적인 힘을 어느정도 가했기 때문에 DNase를 처리 했을 때처럼 다양한 size의 DNA가 관찰 되지만 화학적으로 작용하는 DNAse보다는 좀 불균일하게 size가 분포되었다.





!&emoji=☕&slug=xFu35q9&button_colour=5F7FFF&font_colour=ffffff&font_family=Poppins&outline_colour=000000&coffee_colour=FFDD00)



![[일반생물학실험]온도와 pH가 효소작용에 미치는 영향 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEjdvNA5S925m8lScjFhJj-uy5SdCcaNFG1AuR_7BbQbDEoHBDEEkqfM64sipvro3RydmSO4_MS2794o2bJ0FCljQ31pcD5W39ZGOHROD7qKXLO1rlm2ZuBtbxJ9CZHSnsk2v1KSEsL4EGA/w680/%25EC%2598%25A8%25EB%258F%2584%25EC%2599%2580+pH%25EA%25B0%2580+%25ED%259A%25A8%25EC%2586%258C%25EC%259E%2591%25EC%259A%25A9%25EC%2597%2590+%25EB%25AF%25B8%25EC%25B9%2598%25EB%258A%2594+%25EC%2598%2581%25ED%2596%25A5.PNG)
![[일반생물학실험]여러 조건에 따른 효소 반응](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEj4kaETe9N8j9q9-ClYy9V1kM2L9s5l19K6YoBOE0SB70BmssQXzVADvQijp1ieiQ9mIxcmCwz9e-YCQFVnXYR7m1vmfjTSs0ZNxCmBLG7wgzRBqiY1Kizx8YuhSdw-RDbs1PohokUd69c/w680/%25EC%2597%25AC%25EB%259F%25AC+%25EC%25A1%25B0%25EA%25B1%25B4%25EC%2597%2590+%25EB%2594%25B0%25EB%25A5%25B8+%25ED%259A%25A8%25EC%2586%258C+%25EB%25B0%2598%25EC%259D%2591.PNG)
![[일반생물학실험]항생제 감수성 검사 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEhwCJEjB_r2Ojsrn244q-fQmpHsDyUUfgWMUnsfhPKQeN2e7_u1pN_ZuwIdxPLVWS6LFywJUrDTYWJe2V6ZVVb1PtfyvX62Y15OxPfXoiaJkqVoSir3ZoFAmDpe5GcoMBmxEsJUKsSvWYc/w680/%25ED%2595%25AD%25EC%2583%259D%25EC%25A0%259C+%25EA%25B0%2590%25EC%2588%2598%25EC%2584%25B1+%25EA%25B2%2580%25EC%2582%25AC.PNG)
![[고분자공학실험]PMMA 중합 반응 - MMA, AIBN 정제와 PMMA Bulk 중합 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEj5hkRZhYWcXolP679_yO0kyP8_R-G9dlTqk5AGEdfsIq9M5K3DJc_tvpYck4x1kciBm2IWwFygrzfWH-HPzUjXiCHAFrjv9zeKYDxAl2tGNqgryyc3t9y4B9lQveS_coAqnmZw4pX4Hyo/w680/PMMA+%25EC%25A4%2591%25ED%2595%25A9+%25EB%25B0%2598%25EC%259D%2591.PNG)
![[일반화학실험]산화-환원 적정 : 비타민 C의 정량 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEintFUW1pYHbKK8MPzki4vdqVMjTUEFFxPdZB1sTJQw13XSFZWqcN5w0x1AlyzfErlYYSgoNqBni4YGlpFC4ZeULK9VHbhfIXLiuZ3UWTl2oBRua7FE8UUQqtYEx3498FENAnlHlh0f8RFkT7Na8jveOtBtQ5UbY8jE80ZCwFoU_jqw_0Z6BofRYZF7/w680/%EC%82%B0%ED%99%94-%ED%99%98%EC%9B%90%20%EC%A0%81%EC%A0%95%20-%20%EB%B9%84%ED%83%80%EB%AF%BC%20C%EC%9D%98%20%EC%A0%95%EB%9F%89.PNG)
0 댓글