실험 기구 및 시약
1) DNA template, primer, DW, DNA polymerase & Buffer
2) dNTPs (dATP, dGTP, dCTP, dTTP), PCR tube, micro pipette, column
3) buffer PB, buffer NW, buffer EB, T vector, 2X Rapid Ligation Buffer
4) pGEM®-T or pGEM®-T Easy Vector (50 ng), PCR product, T4 DNA Ligase gel
5) 도말 plate, 따뜻한 물, shaking machine
실험 방법
1) 형광 발현 유전자를 PCR로 cloning한다.
① 튜브에 DNA template 1㎕, FW primer 2㎕, RV primer 2㎕, DW 15㎕를 넣으려고 하였으나 실패의 가능성이 있으므로 각각 2배의 양으로 한다.
② 그 다음 polymerase, buffer들이 있는 PCR tube에 옮겨 담은 다음 잘 섞이도록 pipetting을 해준다. PCR tube에는 라벨링을 한다.
→ PCR tube 속에 있는 파란 물질은 염색을 하기 위한 용도로 전기영동 시에 홈에 주입하였는지 확인을 더 쉽게 할 수 있다.
③ 94℃에서 2분 동안 진행하고 94℃에서 30초, 58℃에서 30초, 72℃에서 1분 30초로 한 cycle을 설정하여 35cycle을 진행한다. 그런 후 4℃에서 보관한다.
→ cycle이나 온도, 시간의 설정은 충분히 primer가 붙을 수 있도록 설정한다.
④ 2㎕을 전기영동한다.
2) PCR 산물을 분리, 정제한다.
① PCR 산물의 볼륨의 5배에 해당하는 양의 buffer PB를 넣은 후에 준비된 column에 모두 옮겨 담고 30초 동안 원심분리 한다.
② 아래쪽에 필터링 된 물질을 pipette으로 제거한 후 buffer NW를 700㎕ 추가한 후에 30초 동안 원심분리 한다. 원심분리 후에는 필터링 된 물질을 제거한다.
③ 1분 동안 추가적으로 원심분리를 하고난 후에 새로운 1.5㎖ tube로 column을 옮긴다.
④ 30㎕의 buffer EB를 넣고 1분 동안 원심분리를 한다.
⑤ 1.5㎖ tube에 있는 물질이 정제된 PCR산물.
⑥ 2㎕을 전기영동한다.
3) 2X Rapid Ligation Buffer - 5 ㎕, pGEM®-T Easy Vector (50 ng) - 1 ㎕, PCR product - 3 ㎕, T4 DNA Ligase - 1 ㎕로 총 10㎕를 튜브에 넣고 이 튜브를 1시간 동안 실온에 보관한다.
4) 칼슘을 처리한 박테리아가 해동될 때 까지 얼음에 보관하고 이후 조심스럽게 세포를 반응 튜브 안으로 주입한다.
5) 튜브를 얼음에 30초 동안 보관한다,
6) 42℃의 물에 90초 동안 놔두었다가 이후 즉시 2분간 얼음에 다시 놔둔다. → 열처리 시에 벡터가 들어간다.
7) 실온에서 SOC medium 800㎕에 반응한 산물을 넣고 37℃에서 45분 동안 인큐베이터에서 shaking한다.
8) 도말할 gel을 미리 만들어 plate에 부어 굳힌다.
9) transformation된 산물을 인큐베이터 안에서 plate에 도말하여 하루 밤이 지나면 관찰한다. → 실제 관찰은 실험이 끝난 후 5일 뒤 관찰.
실험 결과
1) 전기영동 확인 결과
PCR 산물은 약 1300bp에 가깝다고 예상되며 정제된 PCR 산물 역시 비슷하게 관찰된다. 이는 PCR 산물이 전기영동 된 것과 정제한 PCR 산물의 bp가 비슷함을 보여주는데 전기영동에서 이동하는 이동상은 charge를 띠는 것이기 때문에 정제를 하지 않아도 전기영동으로 얻어지는 PCR 산물의 결과는 정제 후의 PCR 산물과 같을 것으로 예상된다.
2) transformation 확인
왼쪽의 사진은 일반 조명에서 관찰 한 모습이며, 오른쪽의 사진은 UV 조명에서 관찰한 것이다. 각각의 사진에서 왼쪽의 결과물은 transformation이 된 것이고, 오른쪽의 결과물은 유전자 변형이 되지 않은 것이다. transformation이 된 plate에서는 파란색의 colony가 보이는데 이것은 변형유전자가 제대로 삽입이 되지 않은 것이다.
원래 실험결과에서의 blue colony : white colony = 1:100 정도라고 한다. 실험이 잘 되었기 때문에 파란 colony의 수는 많지 않았다. 일반 조명아래에서 관찰하였을 때에는 하얀 colony간의 차이가 없지만 UV조명에서 관찰하면 확실한 차이를 알 수 있다. T vector에 변형한 유전자를 삽입하였기 때문에 그 조작된 유전자가 발현되어 이러한 결과가 나타나는 것이다. 각각의 튜브에는 plate에서 하나의 colony를 배양한 것으로 각 사진의 왼쪽은 변형된 colony, 오른쪽은 변형되지 않은 colony이다.
토의 사항
실험에 사용된 T-vector는 운반체라고 할 수는 있지만 그 용도는 클로닝이 주 목적이다. 다른 클로닝 벡터와 T-vector의 차이는 다른 벡터의 경우에는 클로닝은 제한효소 절단 부위를 맞추어 주는 것이 대부분이다. 즉, 벡터에 삽입될 효소절단부위와 삽입되는 DNA조각이 같은 효소절단부위로 이루어져야한다. 그래서 백터가 BamH1으로 절단되면 삽입될 DNA도 BamH1이 사용되어야 합니다. 물론 예외도 있다. 그러나 T 벡터는 순전히 PCR 증폭산물을 클로닝하기 위하여 개발된 벡터이다.
PCR 증폭산물은 제한효소와 관계없이 한결같이 같은 DNA 말단부위가 나온다. PCR에 사용되는 효소인 Taq DNA polymerase는 주형 DNA를 복제하는데 꼭 마지막에 주형에는 없는 A를 첨가한다. (이것을 A overhang이라고 한다.) 따라서 끝이 뭉툭하게 되어야 할 것 같은 DNA 증폭산물이 끝에 A가 나와서 일반 평활말단 벡터에는 삽입이 되지 않는다. 따라서 이 PCR 증폭산물을 클로닝하기 위하여 개발된 것이 T-vector이다. 당연히 클로닝 부위는 T가 튀어나와서 PCR산물의 A와 결합되게 된다.
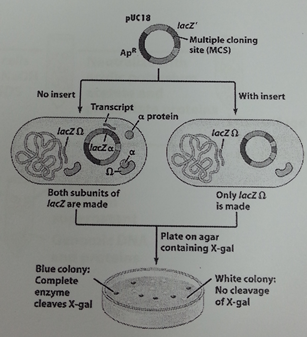
벡터에 GFP가 들어가면 lacZ'가 부서지게 되고 우리가 넣은 것은 lacZ에서 발현이 되지 않는다. 하지만 GFP가 제대로 들어가지 않아서 lacZ가 그대로인 경우에는 lacZ의 두 개의 소단위체가 만들어져서 X-gal의 절단이 일어나지 않아 흰색을 나타내낸다. GFP가 들어간 colony는 lacZΩ만 만들어져서 X-gal의 절단이 완벽하게 일어나서 형광발현이 되어 파란색으로 보이게 된다. 또한 실험에서 사용된 형광단백질은 해파리에서 추출한 형광단백질을 사용한다.





!&emoji=☕&slug=xFu35q9&button_colour=5F7FFF&font_colour=ffffff&font_family=Poppins&outline_colour=000000&coffee_colour=FFDD00)



![[일반생물학실험]온도와 pH가 효소작용에 미치는 영향 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEjdvNA5S925m8lScjFhJj-uy5SdCcaNFG1AuR_7BbQbDEoHBDEEkqfM64sipvro3RydmSO4_MS2794o2bJ0FCljQ31pcD5W39ZGOHROD7qKXLO1rlm2ZuBtbxJ9CZHSnsk2v1KSEsL4EGA/w680/%25EC%2598%25A8%25EB%258F%2584%25EC%2599%2580+pH%25EA%25B0%2580+%25ED%259A%25A8%25EC%2586%258C%25EC%259E%2591%25EC%259A%25A9%25EC%2597%2590+%25EB%25AF%25B8%25EC%25B9%2598%25EB%258A%2594+%25EC%2598%2581%25ED%2596%25A5.PNG)
![[일반생물학실험]여러 조건에 따른 효소 반응](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEj4kaETe9N8j9q9-ClYy9V1kM2L9s5l19K6YoBOE0SB70BmssQXzVADvQijp1ieiQ9mIxcmCwz9e-YCQFVnXYR7m1vmfjTSs0ZNxCmBLG7wgzRBqiY1Kizx8YuhSdw-RDbs1PohokUd69c/w680/%25EC%2597%25AC%25EB%259F%25AC+%25EC%25A1%25B0%25EA%25B1%25B4%25EC%2597%2590+%25EB%2594%25B0%25EB%25A5%25B8+%25ED%259A%25A8%25EC%2586%258C+%25EB%25B0%2598%25EC%259D%2591.PNG)
![[일반생물학실험]항생제 감수성 검사 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEhwCJEjB_r2Ojsrn244q-fQmpHsDyUUfgWMUnsfhPKQeN2e7_u1pN_ZuwIdxPLVWS6LFywJUrDTYWJe2V6ZVVb1PtfyvX62Y15OxPfXoiaJkqVoSir3ZoFAmDpe5GcoMBmxEsJUKsSvWYc/w680/%25ED%2595%25AD%25EC%2583%259D%25EC%25A0%259C+%25EA%25B0%2590%25EC%2588%2598%25EC%2584%25B1+%25EA%25B2%2580%25EC%2582%25AC.PNG)
![[고분자공학실험]PMMA 중합 반응 - MMA, AIBN 정제와 PMMA Bulk 중합 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEj5hkRZhYWcXolP679_yO0kyP8_R-G9dlTqk5AGEdfsIq9M5K3DJc_tvpYck4x1kciBm2IWwFygrzfWH-HPzUjXiCHAFrjv9zeKYDxAl2tGNqgryyc3t9y4B9lQveS_coAqnmZw4pX4Hyo/w680/PMMA+%25EC%25A4%2591%25ED%2595%25A9+%25EB%25B0%2598%25EC%259D%2591.PNG)
![[일반화학실험]산화-환원 적정 : 비타민 C의 정량 1부](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEintFUW1pYHbKK8MPzki4vdqVMjTUEFFxPdZB1sTJQw13XSFZWqcN5w0x1AlyzfErlYYSgoNqBni4YGlpFC4ZeULK9VHbhfIXLiuZ3UWTl2oBRua7FE8UUQqtYEx3498FENAnlHlh0f8RFkT7Na8jveOtBtQ5UbY8jE80ZCwFoU_jqw_0Z6BofRYZF7/w680/%EC%82%B0%ED%99%94-%ED%99%98%EC%9B%90%20%EC%A0%81%EC%A0%95%20-%20%EB%B9%84%ED%83%80%EB%AF%BC%20C%EC%9D%98%20%EC%A0%95%EB%9F%89.PNG)
0 댓글